Regressão Logística usando Tidymodels
By Lucas Moraes in regressão logística tidymodels roc
January 27, 2022
O objetivo desse post é passar rapidamente por um fluxo de trabalho usando o
{tidymodels} para executar uma regressão logística, comparando-a rapidamente com outro modelo. Não abordei a seleção de features ou a interpretação dos coeficientes, nem realizei uma análise exploratória dos dados.
O {tidymodels} compreende um workflow muito prático para trabalhar com modelagem, sendo relativamente simples analisar a performance e comparar diferentes modelos.
Aqui estou apenas raspando a superfície do que é possível fazer e recomendo fortemente a leitura do livro Tidy modelling with R, que além de excelente para explicar o framework em si, também cumpre a função de pincelar diversos conceitos importantes de modelagem.
Leitura dos dados
Primeiro vou carregar as dependências necessárias para a análise. Aqui vou usar principalmente dois conjuntos de pacotes, o
{tidyverse} e o
{tidymodels}. Adicionalmente, estou usando o pacote
{conflicted}, para lidar com conflitos entre algumas dependências:
library(tidymodels)
library(tidyverse)
library(conflicted)
Em seguida leio a tabela, que também está disponível aqui e trato minimamente as variáveis:
conflict_prefer("spec", "yardstick") # resolvendo conflitos de pacotes
tidymodels::tidymodels_prefer() # preferindo funções do tidymodels
# leitura da tabela
# converti algumas colunas para fator
# omiti os NA
df_heart <-
read.csv("https://www.dropbox.com/s/t1158k6mghefmmn/framingham.csv?dl=1") %>%
mutate_at(c("male","education","currentSmoker",
"prevalentStroke","prevalentHyp","diabetes",
"TenYearCHD"), as.factor) %>%
na.omit()
Com isso tenho uma tabela com as variáveis abaixo:
glimpse(df_heart)
## Rows: 3,656
## Columns: 16
## $ male <fct> 1, 0, 1, 0, 0, 0, 0, 0, 1, 1, 0, 0, 1, 0, 0, 1, 0, 0, ~
## $ age <int> 39, 46, 48, 61, 46, 43, 63, 45, 52, 43, 50, 43, 46, 41~
## $ education <fct> 4, 2, 1, 3, 3, 2, 1, 2, 1, 1, 1, 2, 1, 3, 2, 3, 2, 2, ~
## $ currentSmoker <fct> 0, 0, 1, 1, 1, 0, 0, 1, 0, 1, 0, 0, 1, 0, 1, 1, 1, 1, ~
## $ cigsPerDay <int> 0, 0, 20, 30, 23, 0, 0, 20, 0, 30, 0, 0, 15, 0, 20, 10~
## $ BPMeds <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 1, 0, 0, 0, 0, ~
## $ prevalentStroke <fct> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, ~
## $ prevalentHyp <fct> 0, 0, 0, 1, 0, 1, 0, 0, 1, 1, 0, 0, 1, 1, 1, 1, 0, 0, ~
## $ diabetes <fct> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, ~
## $ totChol <int> 195, 250, 245, 225, 285, 228, 205, 313, 260, 225, 254,~
## $ sysBP <dbl> 106.0, 121.0, 127.5, 150.0, 130.0, 180.0, 138.0, 100.0~
## $ diaBP <dbl> 70.0, 81.0, 80.0, 95.0, 84.0, 110.0, 71.0, 71.0, 89.0,~
## $ BMI <dbl> 26.97, 28.73, 25.34, 28.58, 23.10, 30.30, 33.11, 21.68~
## $ heartRate <int> 80, 95, 75, 65, 85, 77, 60, 79, 76, 93, 75, 72, 98, 65~
## $ glucose <int> 77, 76, 70, 103, 85, 99, 85, 78, 79, 88, 76, 61, 64, 8~
## $ TenYearCHD <fct> 0, 0, 0, 1, 0, 0, 1, 0, 0, 0, 0, 0, 0, 0, 1, 0, 1, 0, ~
Estes dados se referem à dados coletados de pacientes em um estudo que procura entender que fatores podem estar ligados à problemas cardíacos futuros (mais detalhes na
página do Kaggle referente a este dataset). A variável de resposta nesse caso vai ser a coluna TenYearCHD.
Partições de treino, teste & replicatas
A primeira coisa a ser feita é particionar os dados. Basicamente quero três conjuntos de dados:
- Um conjunto de dados de treino.
- Um conjunto de dados de teste.
- Um conjunto de replicatas para testar a performance do modelo.
Para executar os primeiros dois passos acima, vou utilizar as funções abaixo:
set.seed(123)
heart_split <-
initial_split(df_heart, prop = 0.80)
A função initial_split cria uma variável que armazena informações sobre as partições de treino, teste e conjunto completo de dados, a partir da tabela df_heart. O argumento prop indica em que proporção minha tabela original será particionada.
O tamanho de cada partição podem ser conferidos chamando a variável:
heart_split
## <Analysis/Assess/Total>
## <2924/732/3656>
Desta variável, extraio as tabelas de treino e teste executando as funções abaixo:
heart_train <- training(heart_split)
heart_test <- testing(heart_split)
A variável heart_train contém os dados de teste, que compõe 80% do total da amostra. O restante encontra-se na variável heart_test. Toda essa separação ajuda a evitar o vazamento de dados de modelagem entre as partições, o que prejudicaria o processo.
Por último, vou criar um conjunto de dados com replicatas dos dados de treino, reamostrados entre si:
set.seed(123)
heart_folds <- vfold_cv(heart_train)
No caso acima utilizei uma validação cruzada para criar as replicatas.
Estas serão utilizadas para testar a performance do modelo ajustado.
Criando receitas
Agora é hora de criar a “receita” da modelagem. Isso envolve basicamente definir qual será a fórmula utilizada na regressão bem como qualquer ajuste a mais que deva ser feitos nos dados:
heart_recipe <-
recipe(TenYearCHD ~ male + age + cigsPerDay + prevalentStroke +
prevalentHyp + totChol + sysBP + glucose,
data = heart_train) %>%
step_center(all_numeric_predictors()) %>%
themis::step_downsample(TenYearCHD)
Esta é uma etapa onde muito ajuste fino pode ser feito. Acima fiz apenas o básico: defini a variáveis de resposta e explicativas e indiquei que todas variáveis descritivas que sejam numéricas deveriam ser centralizadas. Além disso, eu indiquei que deveria ser feito o downsampling nos dados a partir de minha variável de resposta, uma vez que meus dados apresentam desbalanço de classes.
Seguida a esta etapa, defino qual tipo de modelo irei utilizar, neste caso, uma regressão logística:
modelo_log <-
logistic_reg() %>%
set_engine("glm") %>%
set_mode("classification")
Só para deixar as coisas mais interessantes, posso criar uma variável que contém especificações de outro tipo de modelo, como as de uma árvore de decisão:
modelo_dtree <-
decision_tree() %>%
set_engine("rpart") %>%
set_mode("classification")
Dessa maneira, eu posso conferir a performance de cada um dos dois modelos usando a fórmula criada.
Antes de prosseguir, vou definir uma variável que vai servir de argumento para armazenar as previsões feitas nas replicatas:
control <- control_resamples(save_pred = TRUE)
A ideia de usar as replicatas é, em parte, entender como as métricas de performance variam quando ajustamos o modelo em cada uma:
heart_resamples_preds <-
fit_resamples(modelo_log,
heart_recipe,
heart_folds,
control = control)
O que o código acima faz é o seguinte:
- Define qual modelo será ajustado
- Implementa as instruções da receita (fórmula + centralização, no nosso caso)
- Aplica estas etapas nas replicatas, salvando as previsões em tabelas.
A variável heart_resamples_preds contém as informações dos ajustes de cada replicata e, sendo assim, posso extrair uma média das métricas de performance de cada ajuste:
collect_metrics(heart_resamples_preds)
## # A tibble: 2 x 6
## .metric .estimator mean n std_err .config
## <chr> <chr> <dbl> <int> <dbl> <chr>
## 1 accuracy binary 0.660 10 0.00815 Preprocessor1_Model1
## 2 roc_auc binary 0.724 10 0.0151 Preprocessor1_Model1
Na tabela acima, está estimada a acurácia e AUC médias dos ajustes das 10 replicatas acima.
Aqui o interessante é que a sintaxe do tidymodels me permite extrair estas mesmas informações a partir de outro modelo. Dado que criei a variável modelo_dtree, que contém as especificações da árvore de decisões, posso substituir apenas o argumento referente ao modelo sendo ajustado no código e extrair as métricas para este outro algoritmo:
heart_resamples_preds_dtree <-
fit_resamples(modelo_dtree,
heart_recipe,
heart_folds,
control = control)
collect_metrics(heart_resamples_preds_dtree)
## # A tibble: 2 x 6
## .metric .estimator mean n std_err .config
## <chr> <chr> <dbl> <int> <dbl> <chr>
## 1 accuracy binary 0.636 10 0.0101 Preprocessor1_Model1
## 2 roc_auc binary 0.663 10 0.0171 Preprocessor1_Model1
A acurácia foi semelhante à regressão logística, mas a AUC foi bastante inferior. Isto me daria informação o suficiente para prosseguir com a regressão logística em detrimento à árvore de decisões.
O potencial que esta lógica e sintaxe carrega estao no fato de que eu poderia iterativamente testar a performance de \(n\) modelos com os mesmos dados, tratamentos e fórmulas, de maneira bastante simples. Eu poderia, inclusive, testar modelos com fórmulas diferentes, mas isto é tema para outra postagem.
Neste caso específico seguirei com a regressão logística (até porque as métricas parecem indicar que este é o melhor caminho).
Após analisar a AUC das replicatas, o próximo passo é ajustar o modelo na partição de treino, antes disso porém, vou dar uma olhada na curva ROC referente às replicatas:
augment(heart_resamples_preds) %>%
roc_curve(TenYearCHD, .pred_0) %>%
autoplot()
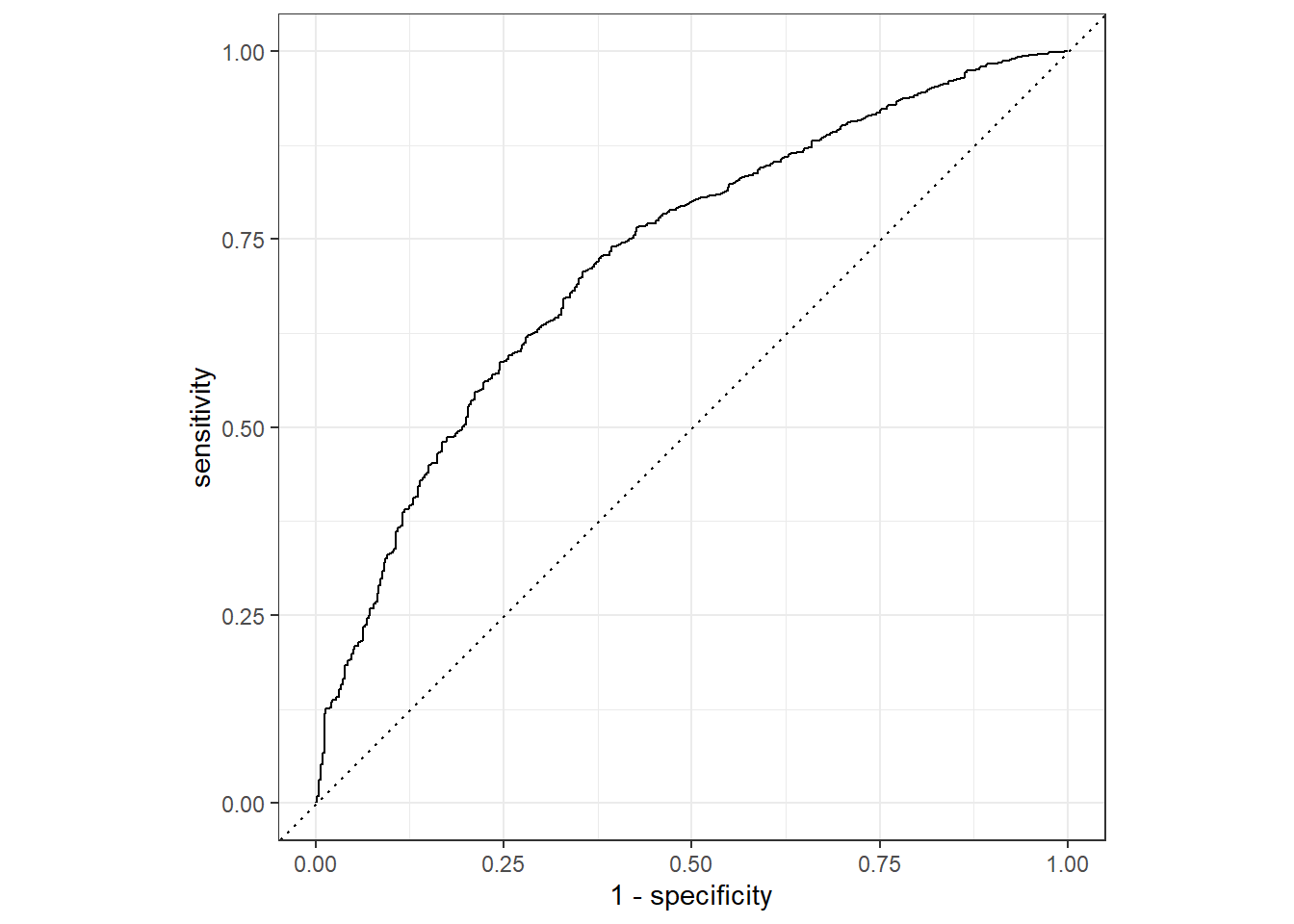
As funções utilizadas acima facilitam a plotagem da curva. Um fato interessante é que o objeto que gera o gráfico é um ggplot e, sendo assim, podemos manipular ele normalmente:
augment(heart_resamples_preds) %>%
roc_curve(TenYearCHD, .pred_0) %>%
autoplot() +
ggtitle("Curva ROC") # adicionando título
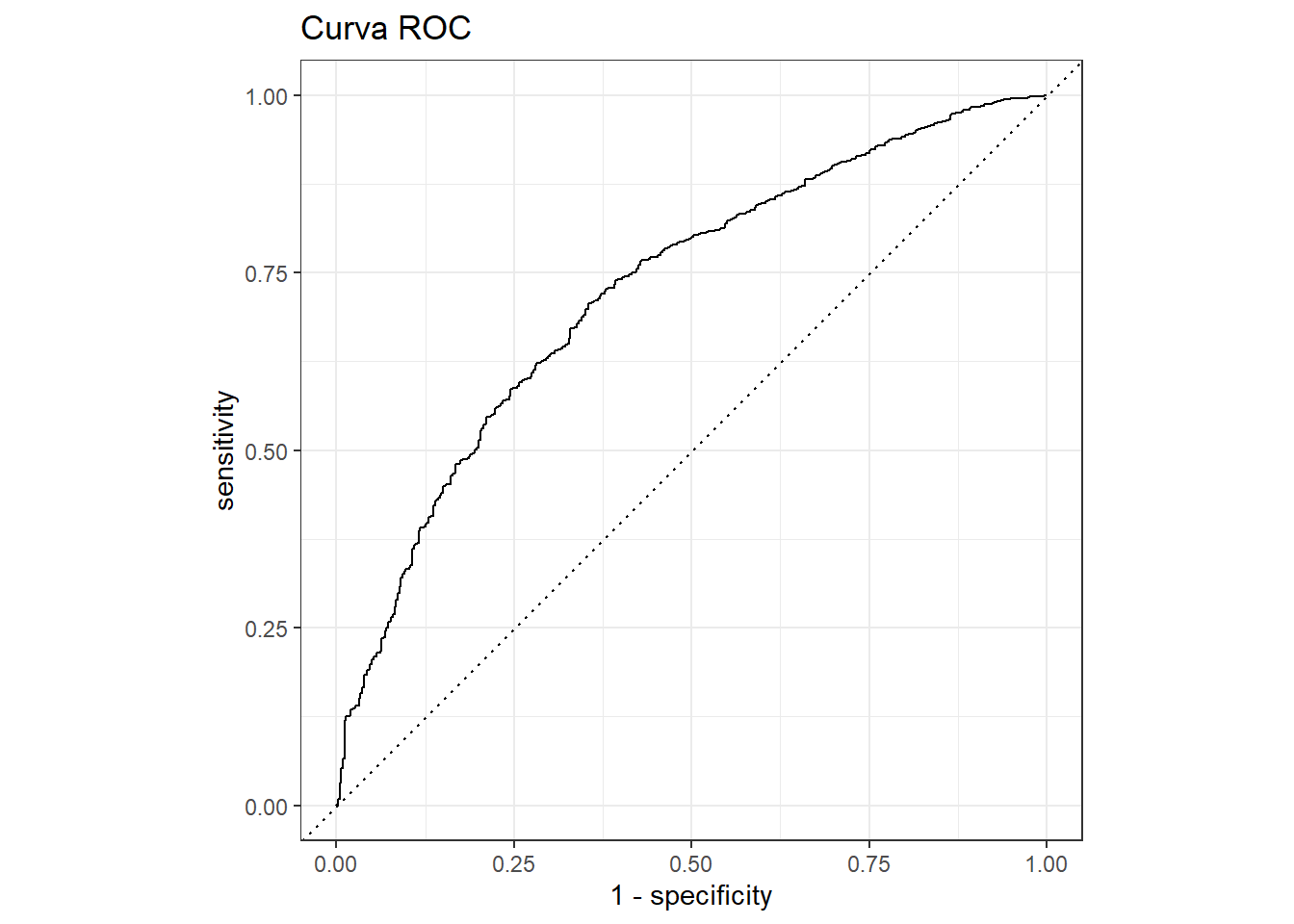
Caso eu quisesse fazer ainda mais ajustes na curva, eu poderia usar o output parcial do código, que gera a tabela utilizada para fazer o plot, que contém os valores de especificidade e sensibilidade:
augment(heart_resamples_preds) %>%
roc_curve(TenYearCHD, .pred_0)
## # A tibble: 2,926 x 3
## .threshold specificity sensitivity
## <dbl> <dbl> <dbl>
## 1 -Inf 0 1
## 2 2.29e-7 0 1
## 3 3.31e-7 0.00227 1
## 4 3.70e-7 0.00455 1
## 5 3.72e-7 0.00455 1.00
## 6 8.25e-7 0.00455 0.999
## 7 3.23e-2 0.00455 0.999
## 8 4.22e-2 0.00682 0.999
## 9 4.88e-2 0.00909 0.999
## 10 5.79e-2 0.0114 0.999
## # ... with 2,916 more rows
Agora, seguirei os mesmos passos acima, mas dessa vez ajustando o modelo à partição de treino:
heart_wflow_fit <-
workflow() %>%
add_model(modelo_log) %>%
add_recipe(heart_recipe) %>%
fit(heart_train)
A partir deste ajuste, vou realizar as previsões na partição de teste. Abaixo estou executando a função predict duas vezes: uma para coletar os outputs na forma de classes e outra na forma de probabilidades:
df_predictions_test <-
heart_test %>%
select(TenYearCHD) %>%
bind_cols(
predict(heart_wflow_fit, heart_test, type = "prob"), # output como probs
predict(heart_wflow_fit, heart_test) # output como classes
)
Vou dar uma conferida em diferentes métricas dessa vez, além da acurácia. Eu posso fazer isso criando uma lista de métricas usando a função metric_set, especificando como argumentos as métricas que quero extrair e, em seguida, usando a função custom_metrics, para gerar o output:
custom_metrics <-
metric_set(accuracy, sens, spec, precision,
recall, f_meas, kap, mcc)
custom_metrics(df_predictions_test,
truth = TenYearCHD,
estimate = .pred_class)
## # A tibble: 8 x 3
## .metric .estimator .estimate
## <chr> <chr> <dbl>
## 1 accuracy binary 0.649
## 2 sens binary 0.631
## 3 spec binary 0.744
## 4 precision binary 0.928
## 5 recall binary 0.631
## 6 f_meas binary 0.751
## 7 kap binary 0.223
## 8 mcc binary 0.277
Abaixo, o valor de AUC e a curva ROC:
roc_auc(df_predictions_test, truth = TenYearCHD,
estimate = .pred_0)
## # A tibble: 1 x 3
## .metric .estimator .estimate
## <chr> <chr> <dbl>
## 1 roc_auc binary 0.752
df_predictions_test %>%
roc_curve(truth = TenYearCHD, .pred_0) %>%
autoplot()
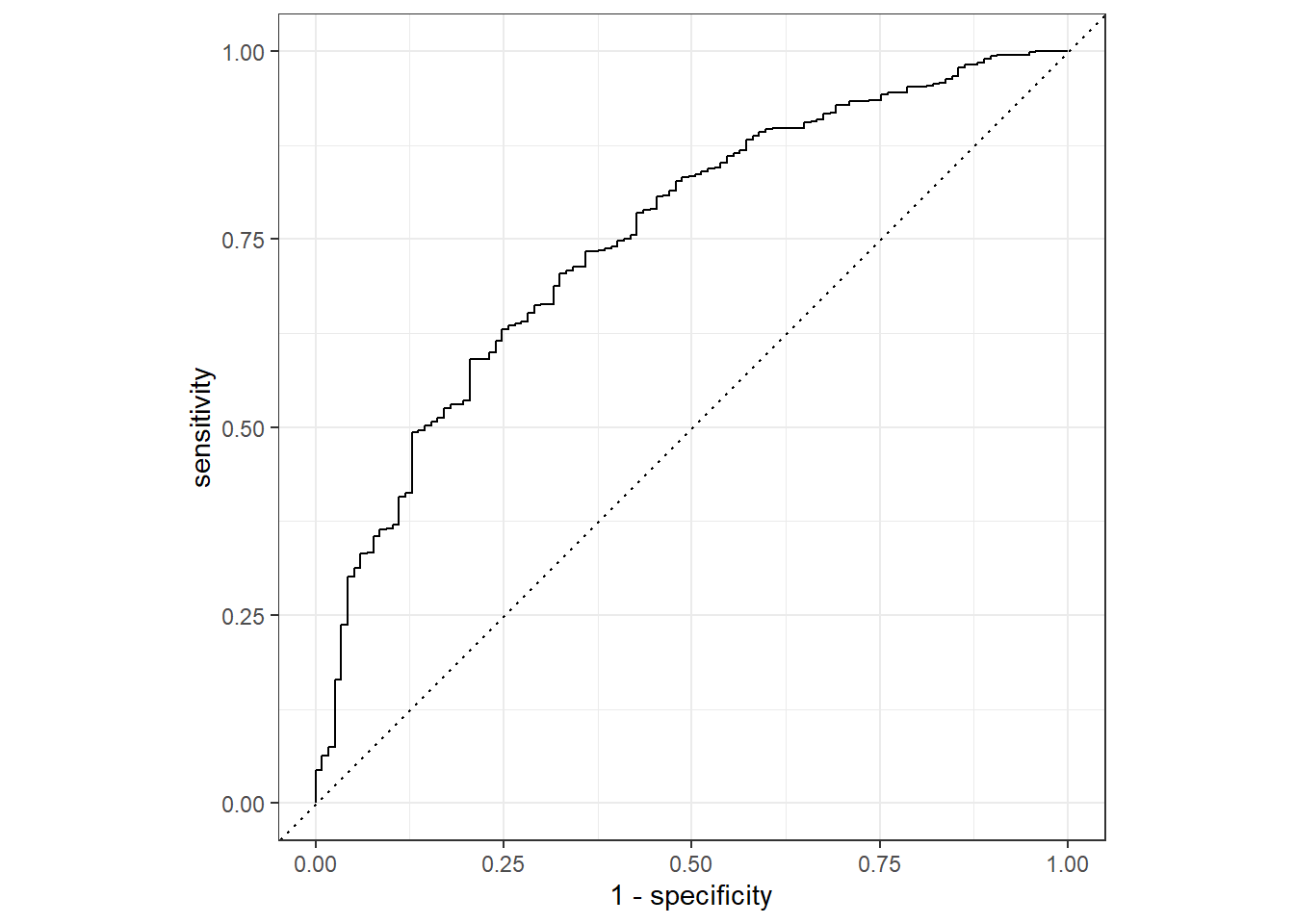
Finalmente, vou conferir os valores dos coeficientes associados ao meu modelo. Posso deixar o output mais amigável e já exponenciado usando a função tidy com o argumento exponentiate = TRUE, aproveitei para deixar a tabela na ordem decrescente de estimativas e filtrei por valores significativos de p:
heart_wflow_fit %>%
tidy(exponentiate = TRUE) %>%
arrange(desc(estimate)) %>%
filter(p.value < 0.05)
## # A tibble: 6 x 5
## term estimate std.error statistic p.value
## <chr> <dbl> <dbl> <dbl> <dbl>
## 1 male1 1.65 0.159 3.17 0.00154
## 2 age 1.06 0.00957 6.04 0.00000000155
## 3 cigsPerDay 1.02 0.00681 2.99 0.00278
## 4 sysBP 1.01 0.00444 2.52 0.0118
## 5 totChol 1.00 0.00170 2.45 0.0145
## 6 (Intercept) 0.556 0.129 -4.53 0.00000578
Conclusões
Tenho me animado recentemente com o uso do {tidymodels} e a motivação deste post foi compartilhar um pouco do porque esse fluxo é interessante e como ele pode aumentar a produtividade na hora de desenvolver modelos.
Abaixo alguns motivos para isso:
-
O
{tidymodels}tem uma boa interface com a sintaxe do{tidyverse}, o que significa um código mais limpo, intepretável, intuititivo e de alto nível. -
As funções já existentes no framework nos permite executar processos com bastante facilidade, sem ter que produzir código específico para isso. No post acima, por exemplo, foi demonstrado como é possível rapidamente comparar a performance de outros modelos com os mesmos dados ou analisar a performance do modelo a partir de replicatas.
-
Interpretar e extrair os resultados das modelagens (valores das previsões e coeficientes, por exemplo) é simples e, em geral, feito de maneira tabular, o que permite uma fácil manipulação dos dados.
-
Fácil plotar a curva ROC!
Existem muitas outras vantagens em se usar esse framework, como o fácil acesso às bibliotecas mais populares de machine learning. Além disso, a estrutura de como o fluxo funciona estimula o uso de boas práticas de modelagem.
- Posted on:
- January 27, 2022
- Length:
- 10 minute read, 2094 words
- Categories:
- regressão logística tidymodels roc
- See Also: